Juliaupを導入してjuliaをバージョンアップさせて、Jupyter labで開発するための備忘録
概要
なんだかんだで二年ぶりの投稿です。
二年間で私の人生も大きく変化してきましたが、直近の経歴はこんな感じです。
- 2023年に某国立大学の博士課程を修了して、博士(工学)を持っています。
- 博士課程在学中に開業してフリーランスとして研究と両立して活動していた経験があり、現在フリーランス4年目です。
- 主に今まで東大スタートアップや大手企業、外資企業など複数の企業でデータサイエンティスト&機械学習エンジニアとして活動しており、推定億単位のレコメンドによる売上改善や特許開発の発明者としての実績があります。最近は機械学習プロジェクトの技術顧問などもしてます。
- 博士課程在学中に平均月収100万円を達成しています(こちらについては近いうちにブログとして投稿する予定です)。
- 丁度2023年9月末でインターンを含め四年以上した企業(正社員)を退職して、今はフリーランスをしながら大学での共同研究を進めています。
かなり前ですが、開業したての情報はこちらに説明があるので、よかったら参考にしてください。
一年近く民間企業の仕事に時間を取られて放置気味だった研究を再開するために、数値計算で使っていたJuliaの環境構築を改めて見直すことになりました。今まではJulia1.5.3を使っていた(古い!)のですが、流石にバージョンアップが必要だと感じており、でもバージョンアップって怖いので今の1.5.3を残しつつアップデートしました。二年ぶりで記事を書く感覚を取り戻してないので、ウォーミングアップという形で備忘録として書いていきます。
前提
既にpythonなどでjupyter環境が入っていて、かつJuliaの何かしらのバージョンをMacにインストールしている方が前提です(今回の記事だと1.5.3)。
手順
まず、juliaupをインストールします。juliaupはjuliaのバージョン管理ツールで、pyenvやnvmと使い勝手が似ていると思います。今回はjuliaupを導入して、既存のjuliaを残しつつ最新のjuliaをインストールしていきます。
$ brew install juliaup
この時、下記のようにシンボリックリンクについてのエラーが発生する可能性が高いです。
Error: The `brew link` step did not complete successfully The formula built, but is not symlinked into /usr/local Could not symlink bin/julia Target /usr/local/bin/julia already exists. You may want to remove it: rm '/usr/local/bin/julia' To force the link and overwrite all conflicting files: brew link --overwrite juliaup To list all files that would be deleted: brew link --overwrite --dry-run juliaup Possible conflicting files are: /usr/local/bin/julia -> /Applications/Julia-1.5.app/Contents/Resources/julia/bin/julia
リンクを削除して、下記を実行します。
$ rm '/usr/local/bin/julia' $ brew link juliaup Linking /usr/local/Cellar/juliaup/1.11.22... 3 symlinks created.
juliaを叩くと、何も入ってない場合は現時点で1.9.3がインストールされます。
$ julia

次に、]を叩いてパッケージ追加するモードに変えて、下記を実行します。これで、jupyterの実行が可能になりました。
(@v1.9) pkg> add Ijulia
通常ならば、ここでjulia1.9.3のバージョンがjupyterのカーネルに登録されるはずなのですが、試しにjupyter kernelspec listと叩いて確認すると、julia1.9.3は見当たりません。ですので、ビルドして登録させます。
julia> using Pkg julia> Pkg.build("IJulia") Building Conda ─→ `~/.julia/scratchspaces/44cfe95a-1eb2-52ea-b672-e2afdf69b78f/8c86e48c0db1564a1d49548d3515ced5d604c408/build.log` Building IJulia → `~/.julia/scratchspaces/44cfe95a-1eb2-52ea-b672-e2afdf69b78f/47ac8cc196b81001a711f4b2c12c97372338f00c/build.log`
この後に、jupyter kernelspec listを叩いてみます。すると、今までの1.5.3のバージョンにjuliaupで入れた1.9.3が共存する形にできました。あとは、jupyter labといつも通りにターミナルから叩けば解決です。
$ jupyter kernelspec list Available kernels: gophernotes /Users/hogehoge/Library/Jupyter/kernels/gophernotes julia-1.5 /Users/hogehoge/Library/Jupyter/kernels/julia-1.5 julia-1.9 /Users/hogehoge/Library/Jupyter/kernels/julia-1.9 python3 /Users/hogehoge/Library/Jupyter/kernels/python3 python3.8.6 /Users/hogehoge/Library/Jupyter/kernels/python3.8.6 rust /Users/hogehoge/Library/Jupyter/kernels/rust
Organizationのリポジトリをgit cloneするときの手順
概要
AWSのEC2環境にssh接続した後に、企業のリポジトリをcloneしようとしたら、うまくいかなかったので解決策を備忘録として書いておきたいと思います。
まず始めに
まずは、環境でsshキーを生成してなければ以下の手順を行いましょう。
- $ cd .ssh
- $ ssh-keygen
- githubのsettingのページに移動して、sshキーを追加する
- .sshフォルダに(作成されてなければ)configファイルを作成して以下を記入する(生成した鍵はid_rsaとしてます)
Host github github.com HostName github.com IdentityFile ~/.ssh/id_rsa User git
gitのユーザー名とメアドの登録
- $ git config --global user.name "ユーザー名"
- $ git config --global user.email "メールアドレス"
Personal tokenを発行してAuthorizeする
非公開のOrganizationのリポジトリをクローンするときは二段階認証が必要なので、以下のQiita記事を参考にしてpersonal access tokenを取得して、private organizationの認証を行います。
二段階認証までやってもcloneできないケースがある場合
vscodeのターミナルなどからクローンするときは、上記の設定をしてもうまくいかないと思います。この場合、vscodeのgithubの拡張機能をサインアウトして再度サインすることで解決します。
解決策は以下に載っていました。サイトの通りに行うと、無事にcloneすることができました。 github.com
まとめ
今回は会社の非公開リポジトリをgit cloneするまでに必要な工程を備忘録として掲載しました。以前も似たような対策をした覚えが何となくあるのですが、最近は色んな企業のリポジトリを扱うことが増えて来たので備忘録として書き留めておくことにしました。
ルンゲクッタ法でHodgkins-Huxleyモデルを実装する
概要
神経モデルとして有名なHodgkins-Huxleyモデルを四次のルンゲクッタ法(Runge-Kutta法)で実装します。 以下のサイトでjuliaを使った神経モデルの実装が幅広く網羅されていますが、Hogkins-Huxleyモデルのコードではオイラー法で更新されていた例しかなかったので、それをルンゲクッタ法に改良しました(ソースコードはこちらを参考に描画のライブラリを変えたりルンゲクッタ法を追加してます)。
Hogkins-Huxleyモデルとは
後日追記しますが、以下の記事に記載されている微分方程式を差分法で更新して解くだけで実装できます。
ルンゲクッタ法とオイラー法の違い
常微分方程式の計算の際には、仕事先だろうが研究先だろうが四次のルンゲクッタで計算したことを自明のように進めていきます。それくらいデフォルト・スタンダードになってます。オイラー法を用いることが推奨されない理由としては、
Euler 法は最も簡単に実装することができる反面、Taylor 展開の 1 次までしか考慮しないため精度がよく ない。
同じ計算を何度も繰り返すため、初期値の点から離れるほど誤差が大きくなってしまう
よって、更新する時間分解能が小さい場合には、適切な更新ができなくなったり、場合によっては異なる挙動を示してしまうことがあります。ルンゲクッタ法では非線形に対応してるため、上記の問題を改善することができます。
具体的な理論や式の導出方法は以下の記事などに詳しく載ってるので説明は省いて、早速実装の方に進んでいきます。
ソースコード
一応、オイラー法とルンゲクッタ法の両方を準備しました。
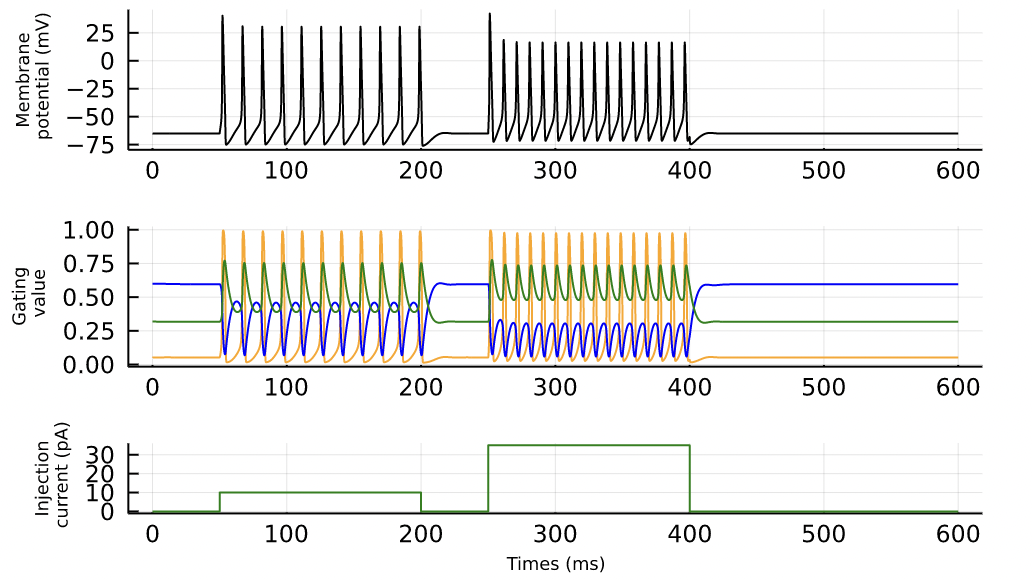
# ライブラリの読み込み using Base: @kwdef using Parameters: @unpack using Plots # Hogkins-Huxleyモデルの定義 @kwdef struct HHParameter{FT} Cm::FT = 1.0 gNa::FT = 120.0 gK::FT = 36.0 gL::FT = 0.3 ENa::FT = 50.0 EK::FT = -77.0 EL::FT = -54.387 tr::FT = 0.5 # ms td::FT = 8.0 # ms invtr::FT = 1.0 / tr invtd::FT = 1.0 / td v0::FT = -20.0 # mV end @kwdef mutable struct HH{FT} param::HHParameter = HHParameter{FT}() N::UInt16 v::Vector{FT} = fill(-65.0, N) m::Vector{FT} = fill(0.05, N) h::Vector{FT} = fill(0.6, N) n::Vector{FT} = fill(0.32, N) r::Vector{FT} = zeros(N) end # オイラー法による更新 function euler_update!(variable::HH, param::HHParameter, Ie::Vector, dt) @unpack N, v, m, h, n, r = variable @unpack Cm, gNa, gK, gL, ENa, EK, EL, tr, td, invtr, invtd, v0 = param @inbounds for i = 1:N m[i] += dt * ((0.1(v[i]+40.0)/(1.0 - exp(-0.1(v[i]+40.0))))*(1.0 - m[i]) - 4.0exp(-(v[i]+65.0) / 18.0)*m[i]) h[i] += dt * ((0.07exp(-0.05(v[i]+65.0)))*(1.0 - h[i]) - 1.0/(1.0 + exp(-0.1(v[i]+35.0)))*h[i]) n[i] += dt * ((0.01(v[i]+55.0)/(1.0 - exp(-0.1(v[i]+55.0))))*(1.0 - n[i]) - (0.125exp(-0.0125(v[i]+65)))*n[i]) v[i] += dt / Cm * (Ie[i] - gNa * m[i]^3 * h[i] * (v[i] - ENa) - gK * n[i]^4 * (v[i] - EK) - gL * (v[i] - EL)) r[i] += dt * ((invtr - invtd) * (1.0 - r[i])/(1.0 + exp(-v[i] + v0)) - r[i] * invtd) end end # ルンゲクッタ法による更新 function df(param::HHParameter, v, m, h, n, Ie) @unpack Cm, gNa, gK, gL, ENa, EK, EL, tr, td, invtr, invtd, v0 = param dm = ((0.1(v+40.0)/(1.0 - exp(-0.1(v+40.0))))*(1.0 - m) - 4.0exp(-(v+65.0) / 18.0)*m) dh = ((0.07exp(-0.05(v+65.0)))*(1.0 - h) - 1.0/(1.0 + exp(-0.1(v+35.0)))*h) dn = ((0.01(v+55.0)/(1.0 - exp(-0.1(v+55.0))))*(1.0 - n) - (0.125exp(-0.0125(v+65)))*n) dv = (1 / Cm) * (Ie - gNa * m^3 * h * (v - ENa) - gK * n^4 * (v - EK) - gL * (v - EL)) return dm, dh, dn, dv end function rk4(param::HHParameter, func, v, m, h, n, dt, Ie) dm_k1, dh_k1, dn_k1, dv_k1 = dt .* func(param::HHParameter, v, m, h, n, Ie) dm_k2, dh_k2, dn_k2, dv_k2 = dt .* func(param::HHParameter, v+0.5*dv_k1, m+0.5*dm_k1, h+0.5*dh_k1, n+0.5*dn_k1, Ie) dm_k3, dh_k3, dn_k3, dv_k3 = dt .* func(param::HHParameter, v+0.5*dv_k2, m+0.5*dm_k2, h+0.5*dh_k2, n+0.5*dn_k2, Ie) dm_k4, dh_k4, dn_k4, dv_k4 = dt .* func(param::HHParameter, v+dv_k3, m+dm_k3, h+dh_k3, n+dn_k3, Ie) v += (dv_k1 .+ 2*dv_k2 .+ 2*dv_k3 .+ dv_k4) ./ 6 m += (dm_k1 .+ 2*dm_k2 .+ 2*dm_k3 .+ dm_k4) ./ 6 h += (dh_k1 .+ 2*dh_k2 .+ 2*dh_k3 .+ dh_k4) ./ 6 n += (dn_k1 .+ 2*dn_k2 .+ 2*dn_k3 .+ dn_k4) ./ 6 return v,m,h,n end function rk_update!(variable::HH, param::HHParameter, Ie::Vector, dt) @unpack N, v, m, h, n, r = variable @inbounds for i = 1:N result = rk4(param::HHParameter, df, v[i], m[i], h[i], n[i], dt, Ie[i]) v[i] = result[1] m[i] = result[2] h[i] = result[3] n[i] = result[4] end end ##################### # メインループ T = 600 # ms dt = 0.01 # ms nt = Int(T/dt) # number of timesteps N = 1 # ニューロンの数 # 入力刺激 t = (1:nt)*dt Ie = repeat(10f0 * ((t .> 50) - (t .> 200)) + 35f0 * ((t .> 250) - (t .> 400)), 1, N) # injection current # 記録用 varr, gatearr = zeros(nt, N), zeros(nt, 3, N) # modelの定義 neurons = HH{Float32}(N=N) # simulation @time for i = 1:nt rk_update!(neurons, neurons.param, Ie[i, :], dt) varr[i, :] = neurons.v gatearr[i, :, :] = [neurons.m'; neurons.h'; neurons.n'] # 修正 end # 描画 p1 = plot(t, varr[:, 1], color="black") labellist=["orange" "blue" "green"] p2 = plot(t, gatearr[:, 1, 1], color=labellist[1]) for i in 2:3 p2 = plot!(t, gatearr[:, i, 1], color=labellist[i]) end; p3 = plot(t, Ie[:, 1], color="green") plot(p1, p2, p3, xlabel = ["" "" "Times (ms)"], ylabel= ["Membrane\n potential (mV)" "Recovery\n current (pA)" "Injection\n current (pA)"], layout = grid(3, 1, heights=[0.4, 0.4, 0.2]), guidefont=font(6), legend=false, size=(500,300))
描画結果ですが、無事に実装できました。
dockerでmecab+neologdの環境構築
概要
今まで使っていたdockerfileがあったんだけど、ppa(deadsnake)の問題なのか分からないけど上手くfetchできなくなってきた汗(でもたまに再起動すると上手くいくのは謎でした。原因がDNS問題やno cache, --fix-missingとかでもないので不明...次回に調べよう)
最近はcolabとかにしていたりFlask/Djangoでインタラクティブな検証環境を作っていたけど、jupyterでも欲しいと言われ始めたので新しいmecab+neologdのdocker環境をdocker-composeでシンプルに構築しました。
参考にしたのはこちら。今回はjupyter labで検証できるようにしたかったので、環境変数とか少しだけ弄ってたりします。また、pythonで使用するライブラリでgccが必要だったので、dockerfileも少し追加しています。
実際の構築環境
作業ディレクトリに以下を配置する
ディレクトリ(想定) ├─ Dockerfile ├─ requirements.txt └─ docker-compose.yml
dockerfileはこちら:requirements.txtに書かれているライブラリをインストールする前に、RUN apt install -y build-essential でgccをインストールしています。
FROM jupyter/base-notebook USER root RUN apt-get update && apt-get install -y \ make \ curl \ file \ git \ libmecab-dev \ mecab \ mecab-ipadic-utf8 RUN git clone --depth 1 https://github.com/neologd/mecab-ipadic-neologd.git RUN mecab-ipadic-neologd/bin/install-mecab-ipadic-neologd -y COPY requirements.txt $PWD RUN apt install -y build-essential RUN pip install -r requirements.txt
docker-compose.ymlがこちら:
version: "3.7"
services:
jupyter-notebook:
image: base-notebook
container_name: test-notebook
build:
context: .
dockerfile: Dockerfile
ports:
- "10000:8888"
environment:
- JUPYTER_ENABLE_LAB=yes
- TZ=Asia/Tokyo
volumes:
- ./work:/home/jovyan/work
構築手順
構築手順は以下の4つだけで、とてもシンプルです。
docker-compose up -d --buildを実行する
docker exec -it <container-ID> /bin/bashで入った後に、jupyter server listでtokenを確認
http://localhost:10000/にアクセスして、tokenを入力して、workディレクトリ以下にあるver1.ipynbを操作する
mecab+neologdは以下で起動します。
import MeCab mecab = MeCab.Tagger("-Owakati -d /usr/lib/aarch64-linux-gnu/mecab/dic/mecab-ipadic-neologd -u /home/jovyan/work/log_words/log_words.dic")
ちなみに、カスタム辞書を作成したりするときはmecab-dict-indexなどが必要ですが、そのパスは
find /usr/ -name mecab-dict-index
で簡単に見つけられますので、初心者の方は覚えておいてください(neologdも同様に探せます)
Juliaでプロットするときに右側のy軸がはみ出ててしまったときの対処法
概要
最近、ジャーナルもアクセプトされたり新規の業務委託の内定を頂いたりと、不安だった出来事が解消されていきました。 二ヶ月以上もブログを更新しなかったのは久しぶりなのですが、今後は少しずつ更新頻度を増やしていきたいと思います。以降しばらくは、ウォーミングアップ的な感じで簡単な内容を載せていきたいと思います。
さて、今回はJuliaでプロットするときに右側のy軸がはみ出してしまう時の対処法です(こちらのissuesを参考にしました)。
例えば、あるsinusoidal inputの発火率に従って、非定常ポアソン過程によって生成されたスパイクと実際の入力をプロットしてみます。すると、右側のy軸がはみ出してしまいました。
dt = 0.01 StopTime=600 t = [0:dt:StopTime;] spike_inds = Tuple.(findall(x -> x > 0, spikes)) # 行列内で0を超えてるスパイクのインデックスの要素を取得 spike_time = first.(spike_inds) # スパイク時系列 neuron_inds = last.(spike_inds) # 神経の番号(1~n_neurons) plot(spike_time, neuron_inds ,st=:scatter,markersize=0.1,color="black") p1 = plot!(twinx(),inputs, color="blue", ylim=(0,600)) plot(p1, xlabel = "Times (ms)", layout = grid(1, 1, heights=[1]), guidefont=font(), legend=false, size=(600,300))

こちらに関してですが、プロットするときにleft_margin, right_marginを設定すれば簡単に対処することができます。ついでにx軸のラベルも少々大きくてはみ出しているので、guidefont=font(8)と設定して修正します。また、ytickfont=font(7)としてy軸の値のフォントの大きさも小さくしてみました。
dt = 0.01 StopTime=600 t = [0:dt:StopTime;] spike_inds = Tuple.(findall(x -> x > 0, spikes)) # 行列内で0を超えてるスパイクのインデックスの要素を取得 spike_time = first.(spike_inds).*dt # スパイク時系列 neuron_inds = last.(spike_inds) # 神経の番号(1~n_neurons) plot(spike_time, neuron_inds ,st=:scatter,markersize=0.1,color="black", ytickfont=font(7)) p1 = plot!(twinx(),t,inputs, color="blue", ylim=(0,400), ytickfont=font(7)) plot(p1, xlabel = "Times (ms)", layout = grid(1, 1, heights=[1]), guidefont=font(8), legend=false, size=(600,300), left_margin = 5Plots.mm, right_margin = 15Plots.mm)
結果は以下の通りです。はみ出すことなく、綺麗にプロットすることができました。

JuliaでBitArray{n}をArray{Bool, n}に変換する方法
スパイク関連のニューラルネットを作っていたりすると特定の条件下でtrue/flaseを1 or 0に変換された配列を使いたくなる時がありますが、例えばiszero.やisone.で要素ごとに生成された乱数を1 or 0で纏めようとすると、BitArrayで出てきてしまいます。
spikes = rand(0:3,100) spikes = isone.(spikes) 結果: 100-element BitArray{1}:
これでも問題ないのですが、個人的にはなるべく配列で関数内部を処理したいことが多いです。例えば以下のように配列で処理したい関数を作ります。
function func_call!(spikes::Vector{Bool}) hogehogeな処理をする end
そして、先程のspikesを入力としてfunc_call!に入れると配列ではないのでmethod errorになってしまいます。
func_call!(spikes) MethodError: no method matching func_call!( ::BitArray{1}) Closest candidates are: func_call!(::Array{Bool,1}) at In[1]:1
BitArrayとArray{Bool}の違いについては以下のstackoverflowにも説明されています。
簡単に説明すると、Arrayはtrue/falseをそれぞれBoolとして格納していて、それぞれがUInt8で表現されています。8ビット=1バイトになりますので、Array{Bool}の場合は格納するためにNバイトの容量が必要になります。
一方で、BitArrayはtrue/falseを一つのビットで表現しており、8つのビットがUInt8に格納される方式になります。従って、Array{Bool}と比較するとN/8バイトしか必要としないメリットがあります。また、ビット操作を行うことができるのも魅力です。
違いをまとめると、以下のようになります。
・Array{Bool}...一つずつの要素をUInt8(1バイト)で表現
・BitArray...一つずつの要素を1ビットで表現したものを8つ集めてUInt8で表現
しかし、なるべくこちらとしては「配列で扱いたい」ので、BitArrayではなくArray{Bool}に変換したいです。変換方法についてですが、juliaはcollectで簡単に配列として変換することができます。
spikes = rand(0:3,100) spikes = collect(isone.(spikes)) 結果: 100-element Array{Bool,1}:
まとめ
今回はjuliaでのBitArrayとArray{Bool}の違いを説明して、BitArrayからArray{Bool}に変換する方法を説明しました。
venvで新しくjupyterインストールして動かしたら、ローカルのjupyter環境と競合して全ての開発環境が詰んだ件についての対処法
概要
めっちゃ長いタイトルですが、タイトルの通りです。業務でjupyterでコードが欲しいと頼まれたので、従来のcolabで渡すのがダルくてvenv+requirements.txtで配布してしまいました。そして、地獄はスタートしました。
なぜか、ローカルのjupyter環境が全て動かなくなりました😇
jupyterは起動する。しかし、あらゆるコードが実行できず、*のまま停止する。juliaをターミナルから起動してnotebook()したときは動くのに、なぜか素でjupyter起動すると動かない。しかも、jupyter起動した時のデフォルトのパスがvenv作ったパスからスタートされてしまうバグつき。
もうね、泣きたくなったよ、うん。
さらに、jupyter notebookはコードを開いた瞬間に500エラーを出される始末でした。ぐぅの音も出ない。
ローカルで入れてるjuliaもpythonもRも動かなくて死にそうになりました(休日の開発がオワタ)。それでも、対応していくうちになんとなく原因が分かったのですが、どうやら
「venvで入れたjupyterバージョンとローカルで入れてるjupyterバージョンが競合した」
ことが原因みたいです(えぇ...汗)
そこで、ローカル環境で新しくjupyterを入れようとしたのですが、古かったせいか色んなエラーを吐かれて詰みかけたので、今回は上記のような特殊な経験をしてしまった(ニッチな)人たちの助けとして「新しいjupyterを導入する際に気をつけたいこと」を記載します
解決方法
もしも上の環境を実行していて従来のjupyterが動かない場合は新しいjupyter環境を入れますが、そこそこ古いjupyter環境から新しい環境に変えようとして何も考えずpip install jupyterlabを行うと
ERROR: Cannot uninstall 'terminado'. It is a distutils installed project and thus we cannot accurately determine which files belong to it which would lead to only a partial uninstall.
のようなエラーが吐かれてしまいます。解決策が中々見つからず時間がかかりましたが、
pip install terminado==0.9.1 --user --ignore-installed
を実行すればterminado問題は解決します。しかし、この後にpip install jupyterlabをすると高確率で
ERROR: pip's dependency resolver does not currently take into account all the packages that are installed. This behaviour is the source of the following dependency conflicts. ndex2 3.2.0 requires enum34, which is not installed.
という内容のエラーが出てくると思いますが、これは
pip install enum34
で大丈夫です。ここまできたら、あとは
pip install jupyterlab
で問題なく動いて解決すると思います(無事に開発環境が復旧したので良かった...明日から平日だし焦った...泣
上の対応は主にjupyterlabを最新版に更新するときの障害ですが、venv+requirements.txtにしたせいで思わぬ障害が発生しました汗
まとめ
相手にjupyter形式で環境を提供するときは本当に気を付けましょう(いや、本当に洒落にならないから。めっちゃ謝ることになるから。さっき担当者に謝ったから汗汗汗)
もうトラウマになってしまって、例えどんなに面倒臭くても、二度とvenv+requirements.txtで環境渡せないです笑
余談
ローカルに入れていたjupyter環境とvenv上で新しく入れたjupyter環境の競合が原因なので原因特定に時間がかかりましたが、上記の対応でなんとかなると思います。ちなみに、venv上のカーネルについてはjupyter kernelspec listに登録されていると気持ち悪いと思いますので、
jupyter kernelspec uninstall 仮想環境で作ったカーネル名
で削除できます。あー気持ちいい()