ルンゲクッタ法でHodgkins-Huxleyモデルを実装する
概要
神経モデルとして有名なHodgkins-Huxleyモデルを四次のルンゲクッタ法(Runge-Kutta法)で実装します。 以下のサイトでjuliaを使った神経モデルの実装が幅広く網羅されていますが、Hogkins-Huxleyモデルのコードではオイラー法で更新されていた例しかなかったので、それをルンゲクッタ法に改良しました(ソースコードはこちらを参考に描画のライブラリを変えたりルンゲクッタ法を追加してます)。
Hogkins-Huxleyモデルとは
後日追記しますが、以下の記事に記載されている微分方程式を差分法で更新して解くだけで実装できます。
ルンゲクッタ法とオイラー法の違い
常微分方程式の計算の際には、仕事先だろうが研究先だろうが四次のルンゲクッタで計算したことを自明のように進めていきます。それくらいデフォルト・スタンダードになってます。オイラー法を用いることが推奨されない理由としては、
Euler 法は最も簡単に実装することができる反面、Taylor 展開の 1 次までしか考慮しないため精度がよく ない。
同じ計算を何度も繰り返すため、初期値の点から離れるほど誤差が大きくなってしまう
よって、更新する時間分解能が小さい場合には、適切な更新ができなくなったり、場合によっては異なる挙動を示してしまうことがあります。ルンゲクッタ法では非線形に対応してるため、上記の問題を改善することができます。
具体的な理論や式の導出方法は以下の記事などに詳しく載ってるので説明は省いて、早速実装の方に進んでいきます。
ソースコード
一応、オイラー法とルンゲクッタ法の両方を準備しました。
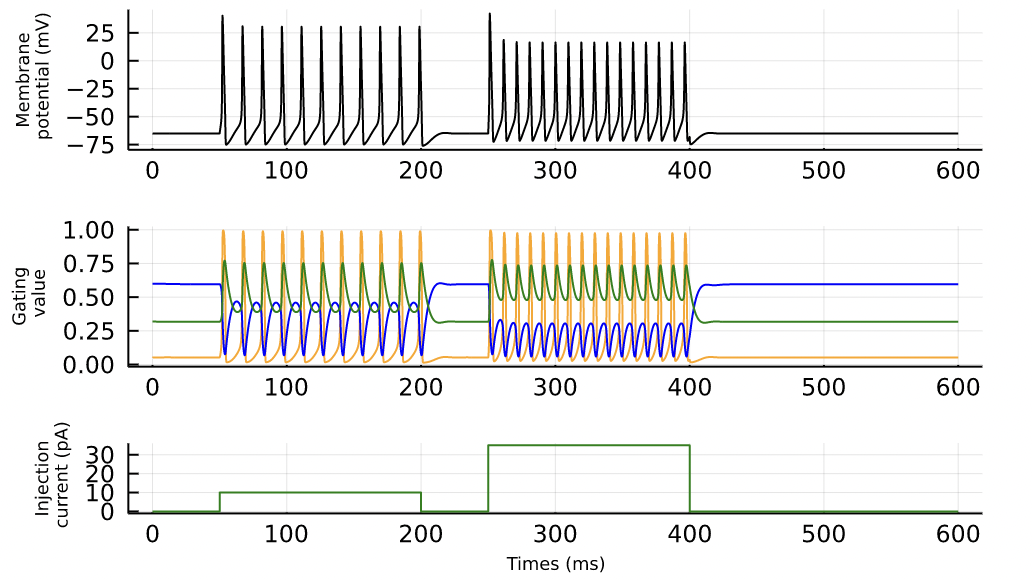
# ライブラリの読み込み using Base: @kwdef using Parameters: @unpack using Plots # Hogkins-Huxleyモデルの定義 @kwdef struct HHParameter{FT} Cm::FT = 1.0 gNa::FT = 120.0 gK::FT = 36.0 gL::FT = 0.3 ENa::FT = 50.0 EK::FT = -77.0 EL::FT = -54.387 tr::FT = 0.5 # ms td::FT = 8.0 # ms invtr::FT = 1.0 / tr invtd::FT = 1.0 / td v0::FT = -20.0 # mV end @kwdef mutable struct HH{FT} param::HHParameter = HHParameter{FT}() N::UInt16 v::Vector{FT} = fill(-65.0, N) m::Vector{FT} = fill(0.05, N) h::Vector{FT} = fill(0.6, N) n::Vector{FT} = fill(0.32, N) r::Vector{FT} = zeros(N) end # オイラー法による更新 function euler_update!(variable::HH, param::HHParameter, Ie::Vector, dt) @unpack N, v, m, h, n, r = variable @unpack Cm, gNa, gK, gL, ENa, EK, EL, tr, td, invtr, invtd, v0 = param @inbounds for i = 1:N m[i] += dt * ((0.1(v[i]+40.0)/(1.0 - exp(-0.1(v[i]+40.0))))*(1.0 - m[i]) - 4.0exp(-(v[i]+65.0) / 18.0)*m[i]) h[i] += dt * ((0.07exp(-0.05(v[i]+65.0)))*(1.0 - h[i]) - 1.0/(1.0 + exp(-0.1(v[i]+35.0)))*h[i]) n[i] += dt * ((0.01(v[i]+55.0)/(1.0 - exp(-0.1(v[i]+55.0))))*(1.0 - n[i]) - (0.125exp(-0.0125(v[i]+65)))*n[i]) v[i] += dt / Cm * (Ie[i] - gNa * m[i]^3 * h[i] * (v[i] - ENa) - gK * n[i]^4 * (v[i] - EK) - gL * (v[i] - EL)) r[i] += dt * ((invtr - invtd) * (1.0 - r[i])/(1.0 + exp(-v[i] + v0)) - r[i] * invtd) end end # ルンゲクッタ法による更新 function df(param::HHParameter, v, m, h, n, Ie) @unpack Cm, gNa, gK, gL, ENa, EK, EL, tr, td, invtr, invtd, v0 = param dm = ((0.1(v+40.0)/(1.0 - exp(-0.1(v+40.0))))*(1.0 - m) - 4.0exp(-(v+65.0) / 18.0)*m) dh = ((0.07exp(-0.05(v+65.0)))*(1.0 - h) - 1.0/(1.0 + exp(-0.1(v+35.0)))*h) dn = ((0.01(v+55.0)/(1.0 - exp(-0.1(v+55.0))))*(1.0 - n) - (0.125exp(-0.0125(v+65)))*n) dv = (1 / Cm) * (Ie - gNa * m^3 * h * (v - ENa) - gK * n^4 * (v - EK) - gL * (v - EL)) return dm, dh, dn, dv end function rk4(param::HHParameter, func, v, m, h, n, dt, Ie) dm_k1, dh_k1, dn_k1, dv_k1 = dt .* func(param::HHParameter, v, m, h, n, Ie) dm_k2, dh_k2, dn_k2, dv_k2 = dt .* func(param::HHParameter, v+0.5*dv_k1, m+0.5*dm_k1, h+0.5*dh_k1, n+0.5*dn_k1, Ie) dm_k3, dh_k3, dn_k3, dv_k3 = dt .* func(param::HHParameter, v+0.5*dv_k2, m+0.5*dm_k2, h+0.5*dh_k2, n+0.5*dn_k2, Ie) dm_k4, dh_k4, dn_k4, dv_k4 = dt .* func(param::HHParameter, v+dv_k3, m+dm_k3, h+dh_k3, n+dn_k3, Ie) v += (dv_k1 .+ 2*dv_k2 .+ 2*dv_k3 .+ dv_k4) ./ 6 m += (dm_k1 .+ 2*dm_k2 .+ 2*dm_k3 .+ dm_k4) ./ 6 h += (dh_k1 .+ 2*dh_k2 .+ 2*dh_k3 .+ dh_k4) ./ 6 n += (dn_k1 .+ 2*dn_k2 .+ 2*dn_k3 .+ dn_k4) ./ 6 return v,m,h,n end function rk_update!(variable::HH, param::HHParameter, Ie::Vector, dt) @unpack N, v, m, h, n, r = variable @inbounds for i = 1:N result = rk4(param::HHParameter, df, v[i], m[i], h[i], n[i], dt, Ie[i]) v[i] = result[1] m[i] = result[2] h[i] = result[3] n[i] = result[4] end end ##################### # メインループ T = 600 # ms dt = 0.01 # ms nt = Int(T/dt) # number of timesteps N = 1 # ニューロンの数 # 入力刺激 t = (1:nt)*dt Ie = repeat(10f0 * ((t .> 50) - (t .> 200)) + 35f0 * ((t .> 250) - (t .> 400)), 1, N) # injection current # 記録用 varr, gatearr = zeros(nt, N), zeros(nt, 3, N) # modelの定義 neurons = HH{Float32}(N=N) # simulation @time for i = 1:nt rk_update!(neurons, neurons.param, Ie[i, :], dt) varr[i, :] = neurons.v gatearr[i, :, :] = [neurons.m'; neurons.h'; neurons.n'] # 修正 end # 描画 p1 = plot(t, varr[:, 1], color="black") labellist=["orange" "blue" "green"] p2 = plot(t, gatearr[:, 1, 1], color=labellist[1]) for i in 2:3 p2 = plot!(t, gatearr[:, i, 1], color=labellist[i]) end; p3 = plot(t, Ie[:, 1], color="green") plot(p1, p2, p3, xlabel = ["" "" "Times (ms)"], ylabel= ["Membrane\n potential (mV)" "Recovery\n current (pA)" "Injection\n current (pA)"], layout = grid(3, 1, heights=[0.4, 0.4, 0.2]), guidefont=font(6), legend=false, size=(500,300))
描画結果ですが、無事に実装できました。